202204推文 Chloroplot-VX
Chloroplot——更好用的叶绿体基因组圈图可视化工具
简介
在测序技术飞速发展的今天,有越来越多的细胞器基因组被组装注释出来。想要理解基因组结构的复杂性及其独特的结构,手段之一就是利用能够将这些特征描绘出来的可视化工具。这些工具确保了数据具有生物学意义。
本期我们来讲一下叶绿体基因组画图,常见的工具有CPGAVAS2、OGDRAW、Chloroplot等,其中Chloroplot拥有丰富的可选项以及鲜明的色彩选择方案,下面来介绍一下Chloroplot的网页版及windows下使用方法。
网页版使用
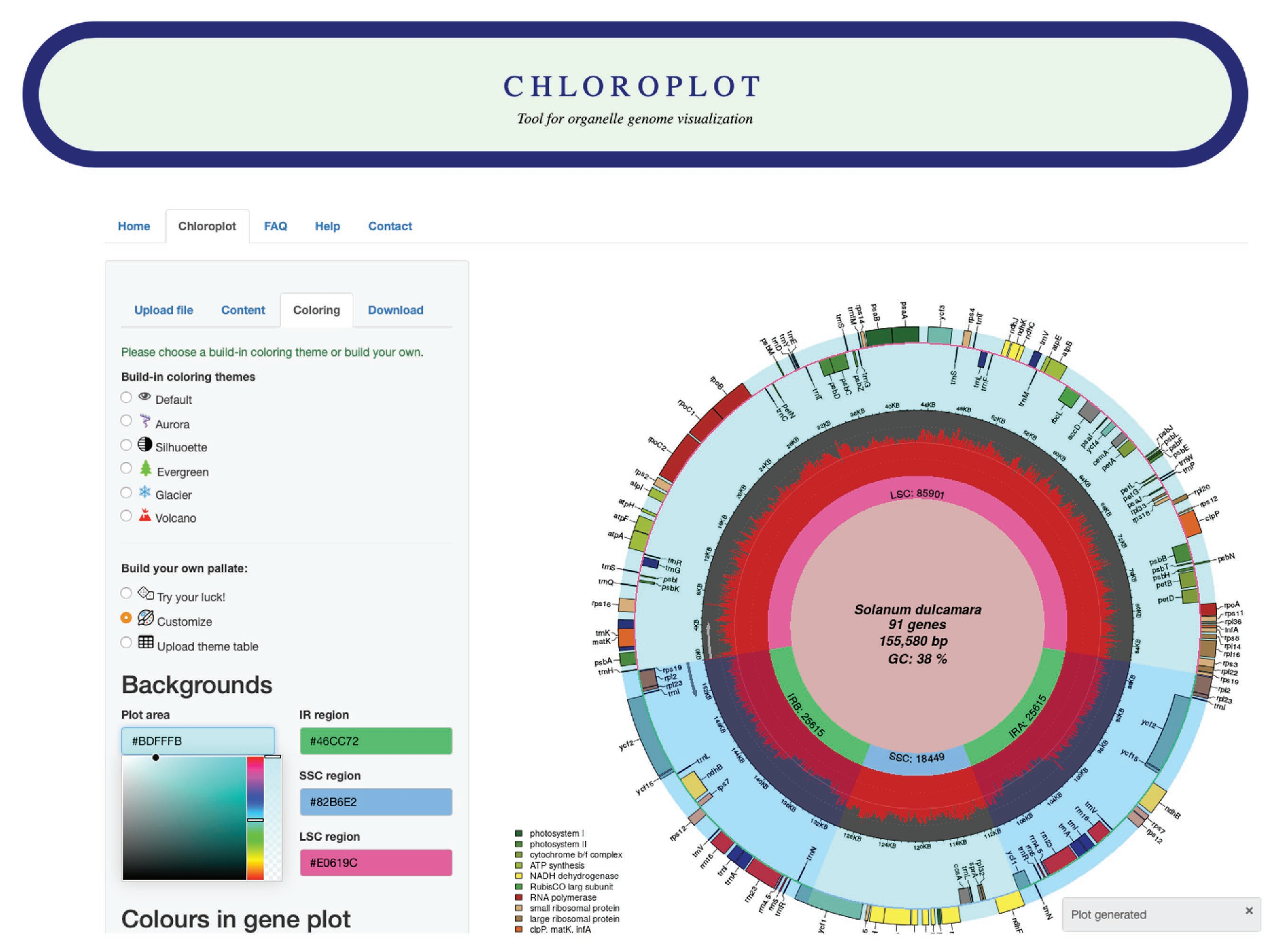
网页版链接https://irscope.shinyapps.io/chloroplot/,主界面如下

使用的时候,点击Chloroplot上传genebank文件,点击plot即可
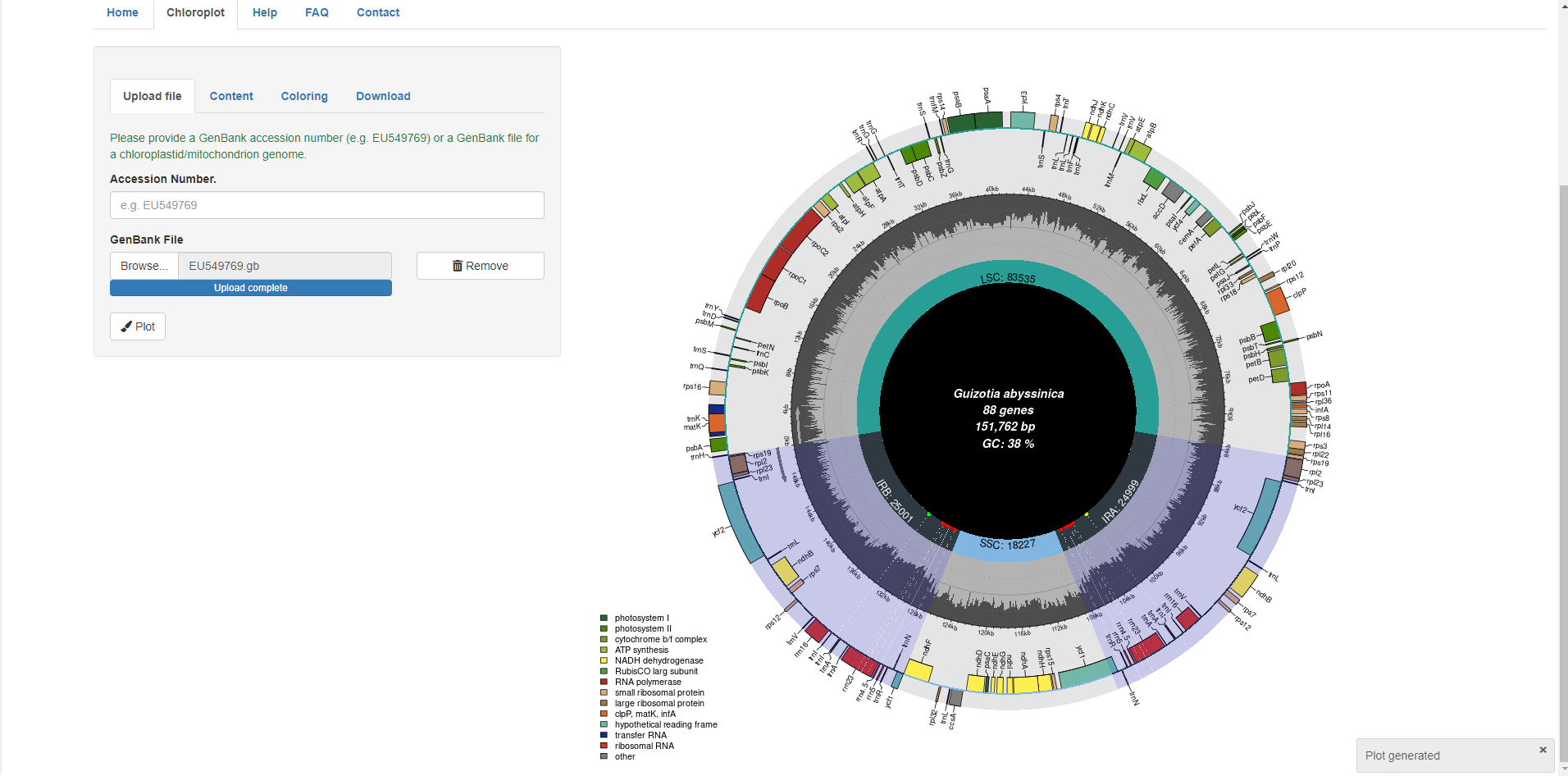
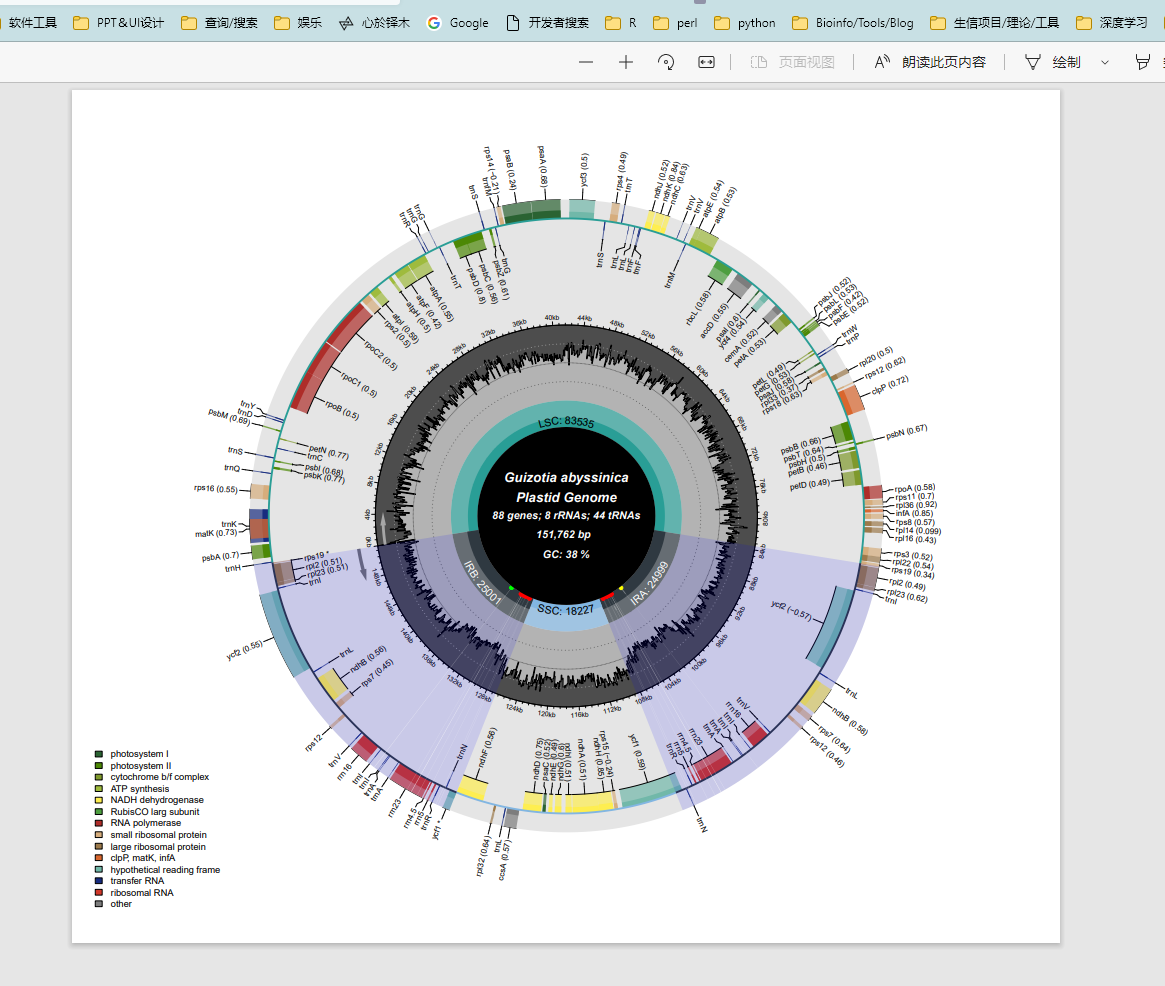
用起来还是很方便的,直接上传文件,就可以获得结果。工具还可以自定义需要展示的内容,如基因组大小、总GC含量、基因的数量、基因组类型(线粒体也是可以画的)、每个基因的GC含量、假基因等。
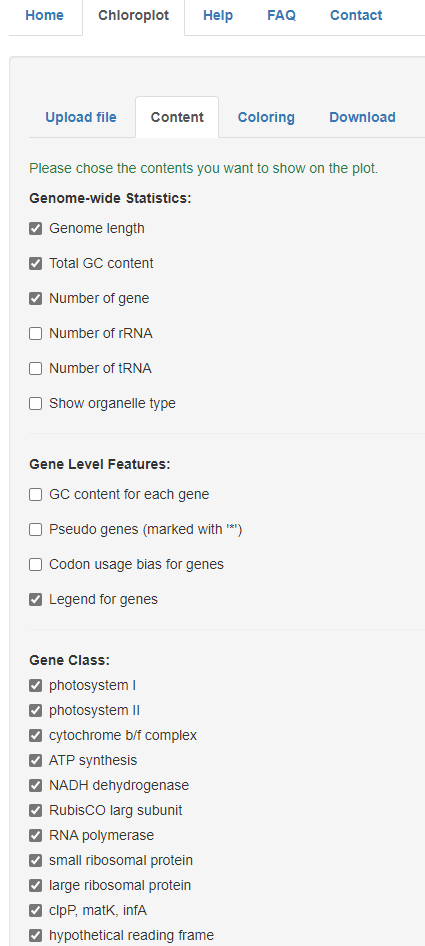
此外,颜色也是可以更改的,拥有非常丰富的色彩方案。每次更改待展示的内容或更改颜色后,工具都会自动重新绘图。最后,画完图记得下载下来,可选文件格式及DPI大小,在此就不多做赘述了。
windows下使用
R 语言是为研究工作者设计的一种数学编程语言,广泛用于统计分析、绘图、数据挖掘。而这款工具本质上是一款R语言工具包,因此我们可以在本地的windows平台和linux平台上运行它,因linux下调试环境比较繁琐及篇幅有限,只讲一下windows下的使用。
1.下载Chloroplothttps://irscope.shinyapps.io/Chloroplot/_w_5ff7b177/Chloroplot.zip,解压缩至本地,例如我的本地路径D:\OneDrive - cancer\Bioinfo_analysis\Chloroplot。
2.安装R语言https://cran.r-project.org/bin/windows/base/R-4.1.2-win.exe,下载后默认安装即可。
3.双击桌面上的快捷方式,一般现在都是64位操作系统,所以我们点击x64那个。

4.在打开的R Console中,依次输入以下内容。
- 先输入以下内容安装导入一些包,不然会报错,可以全部复制进去,按回车。
install.packages("BiocManager")
BiocManager::install("genbankr")
BiocManager::install("coRdon")
install.packages("circlize")
library(dplyr)
- 再输入以下内容,一行一行地输入,输入一行按回车再输下一行**(不要输
#后的内容),注意把D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot替换为你自己的路径**,另外注意斜杠的方向(要么是/要么是\\,不要输\)。
rm(list=ls()) #清空所有变量,井号后为注释,不要复制
setwd("E:/R") #此处设置工作目录,假设**已经在E盘下创建了名为R的文件夹**,此处可以写你自己的文件夹位置
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/color_complement.R")
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/converse_ssc.R")
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/detect_ir.R")
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/GC_count.R")
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/gene_color.R")
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/gene_info.R")
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/parse_gb_file.R")
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/plot_genome.R")
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/read_gb_file.R")
source("D:/OneDrive - cancer/Bioinfo_analysis/Chloroplot/R/test_parameters.R")
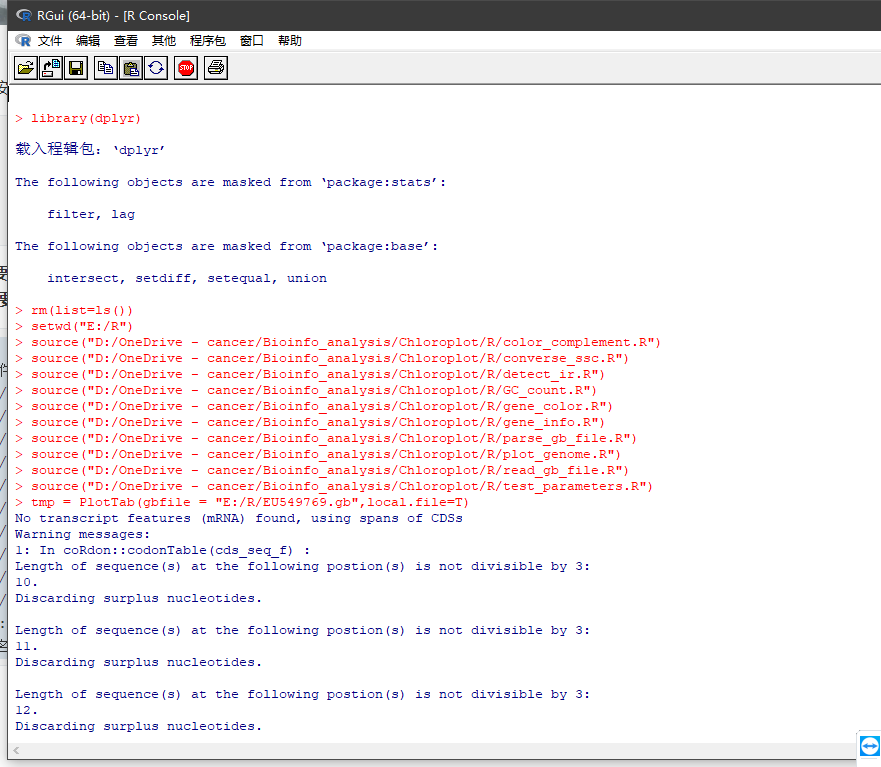
tmp = PlotTab(gbfile = "E:/R/EU549769.gb",local.file=T) #E:/R/EU549769.gb为已经下载的genebank文件位置
PlotPlastidGenome(plot.tables=tmp,file.name="圈图名字")#圈图名字可以自定义,如果不加file.name参数,则生成的pdf与gbk同名
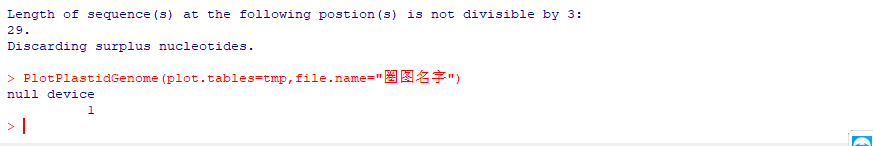
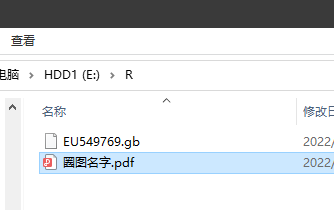
5.我们看到文件夹下已经生成了pdf,可以用浏览器等打开pdf。
后记
1.更多的帮助信息可查看Chloroplot\man文件夹下的.rd文档,里面介绍了更详细的使用方法。
2.Github仓库地址https://github.com/shuyuzheng/Chloroplot。